人工知能技術開発でChIP-Atlasのキュレーション時間を3分の1に短縮
株式会社ヒューマノーム研究所は、株式会社Rhelixa(レリクサ)と共に、九州大学・沖 真弥 助教を中心に開発が行われている転写因子結合サイトのデータベース ChIP-Atlas で実施しているデータキュレーションを半自動化するAIを開発し、本格運用を開始しました。このAI導入により、従来のキュレーション時間を3分の1に短縮することが可能となりました。
ChIP-Atlas は、実験で読まれたDNA配列のデータベースである NCBI Sequence Read Archive (SRA) から、ChIP-seq実験に関するデータをダウンロードし、転写因子結合部位のデータベースとして整理し、可視化やデータマイニングが行えるWebサービスです。このデータベースの一つの特徴として、ChIP-Atlasに収録されたデータ全てに、実験の対象となった転写因子名や細胞名が整然と記述されていることがあげられます。
実はこれ、データを寄託した研究者によって記入された実験のアノテーション情報に対して、全て人力で、ChIP-Atlasチームがキュレーションを実施しています。
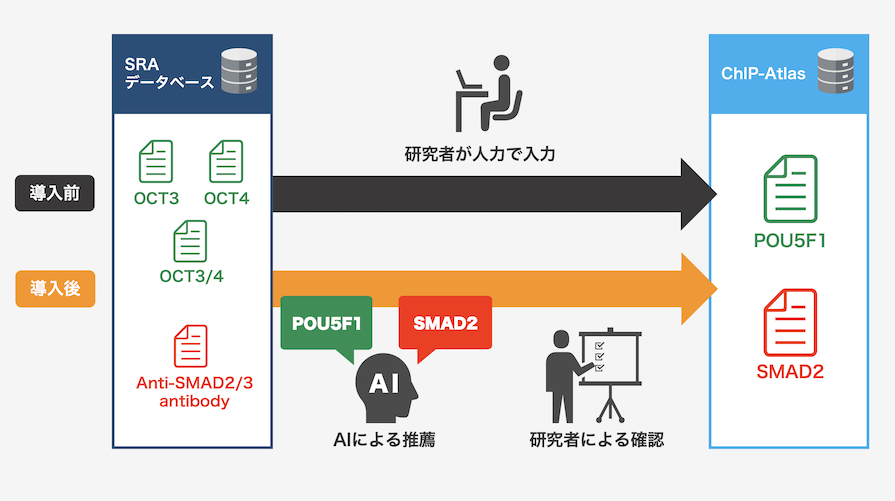
例えば、iPS化の因子として有名な転写因子POU5F1は、OCT3、OCT4、OCT3/4などの別名を持っており、実験登録者は自分の親しんだ名前で登録をします。一方でデータベースのユーザがOCT3で検索を行った場合、同じものであるにも関わらずPOU5F1による実験が見つからない、という現象が起きます。
ChIP-Atlasのキュレータは、これらを全て正式名称のPOU5F1と統一(名寄せ)することで、全データのシームレスな検索を提供しています。細胞名についても同様です。一方で、毎月平均1,500件も登録されるChIP-seqデータについて、この作業を人手で行うことは時間を要する作業であり、ChIP-Atlasを運営する上で大きな手間となっていました。
そこで、ヒューマノーム研究所はレリクサと共同で、過去にChIP-Atlasで行われた10万件近くものキュレーション事例を教師データとして学習し、新たにSRAへ登録された実験のアノテーション情報を自動的にキュレーションするAIシステムを開発いたしました。
これにより、全ての結果をキュレータが修正するのではなく、AIにより推薦された転写因子名や細胞名をキュレータが確認し、問題がなければそのまま採用、もし内容に齟齬があれば人力で修正するというワークフローが確立されました。キュレーション時間は従来の約3分の1と大幅に短縮され、作業の省力化・高速化を実現しています。
研究者間での記載の違いにより情報検索が難しくなる問題は、ChIP-Atlasで利用しているSRAのみならず、カルテなどの記載にも現れる問題です。ヒューマノーム研究所は、今回開発した技術を、今後は医療等におけるデータ整備へと応用していきます。
関連リンク
遺伝子のスイッチ役を「見える化」するシステムについての共著論文を発表しました
株式会社ヒューマノーム研究所
「人間とは何かー」 健康が当たり前な世界において、個々が自分らしく、楽しく暮らせる健康社会を、最先端テクノロジーによるビッグデータの計測と、人工知能(AI)技術の開発・活用を実施することで、人間とそれを取り巻く環境の理解から実現する研究所です。
所在地 | 〒104-0045 東京都中央区築地2-4-10 SAテンハウス2階 |
|---|---|
代表者 | 代表取締役社長 瀬々 潤 |
URL | |
事業内容 | ヒトの理解に関連するAI技術研究、統合解析技術の社会実装 等 |
株式会社Rhelixa(レリクサ)
「生命情報を読み解き、生き物の新たな可能性を創造する」という企業理念のもと、独自のゲノム解析・編集技術により、ゲノムとエピゲノムのデータを繋ぎ、更にはメタゲノムデータを加えた統合的な解析、製品開発を行っています。
本社 | 東京都千代田区岩本町3-7-4 政弥ビル3階 |
|---|---|
代表者 | 代表取締役社長 仲木 竜 |
URL | |
事業内容 | ゲノム・エピゲノムの情報解析およびソフトウェア・装置開発 等 |



